SARS-CoV-2, virusul care de declanșat pandemia actuală și care a ucis aproape 400 de mii de oameni din toată lumea invadează celulele cu ajutorul proteinei “spike” sau proteina S, după cum au explicat oamenii de știință.
Acum, un grup de cercetători de la Universitatea Națională Lehigh și Seul din Coreea de Sud și de la Universitatea Cambridge din Marea Britanie au lucrat împreună pentru a produce primele modele de atomi cu sursă deschisă ale unei proteine S cu lungime întreagă. Cercetătorii spun ca aceasta este de o importanță deosebită, deoarece proteina S joaca un rol central în intrarea virala în celule, ceea ce o face o ținta principala pentru dezvoltarea de vaccinuri și antivirale, transmite Medical Express.
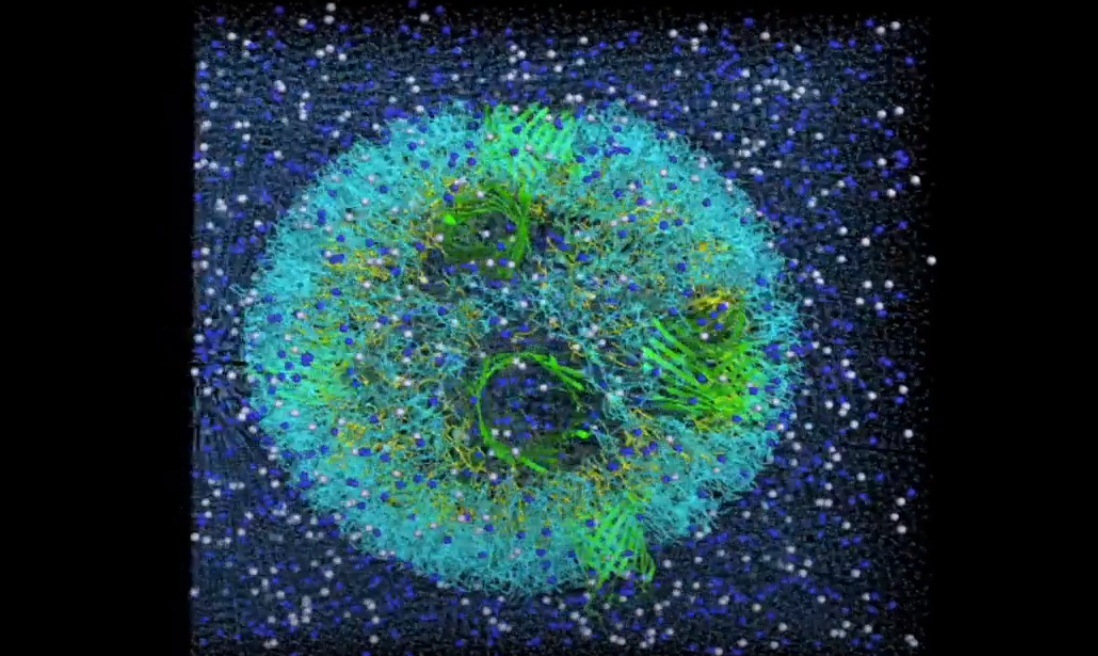
Acest video ilustrează modul de construire a sistemului de membrană din modelele lor de proteine SARS-CoV-2 S. Programul de construire a modelelor are acces deschis și poate fi găsit de pe pagina principală a CHARMM-GUI la Arhiva COVID-19.
Dezvoltat de Wonpil Im, profesor în departamentul de științe biologice și bioinginerie din Lehigh, CHARMM-GUI (interfață grafică de utilizator) este un program care simulează sisteme biomoleculare complexe simplu, precis și rapid. Îl descriu ca un „microscop de calcul” care permite oamenilor de știință să înțeleagă interacțiunile la nivel molecular care nu pot fi observate în alt mod.
„Modelele noastre sunt primele modele de proteine cu SARS-CoV-2 spice (S) complet glicozilate, care sunt disponibile și pentru alți oameni de știință”, spune Im. „Echipa noastră a petrecut zile și nopți pentru a construi aceste modele foarte atent din porțiunile cunoscute ale structurii cryo-EM. Modelarea a reprezentat o provocare mare, deoarece au existat multe regiuni în care modelarea simplă nu a furnizat modele de înaltă calitate. “
Oamenii de știință pot utiliza modelele pentru a efectua cercetări de simulare inovatoare pentru prevenirea și tratamentul COVID-19, potrivit lui Im.
Structura proteinei S a fost determinată cu cryo-EM cu RBD în sus (PDB ID: 6VSB) și cu RBD în jos (PDB ID: 6VXX). Dar acest model are multe reziduuri lipsă. Deci, au modelat mai întâi reziduurile de aminoacizi lipsă, apoi alte domenii lipsă. În plus, au modelat toți glicanii potențiali (sau carbohidrații) atașați la proteina S. Glicanii previn recunoașterea anticorpilor, ceea ce face dificilă dezvoltarea unui vaccin. De asemenea, au construit un sistem de membrană virală a unei proteine S pentru simularea dinamicii moleculare.